醸造微生物学部門の研究紹介
酵母や麹菌は、私たちの日常の食生活に密接に関わっている微生物です。たとえば、パンがふっくら焼き上がるには酵母が必要ですし、ビール・ワイン・日本酒・焼酎などは、酵母の発酵能力によってできあがります。麹菌は、焼酎や日本酒を造るときサツマイモや米などに含まれるデンプンを分解してブドウ糖をつくります。酵母はこのブドウ糖から二酸化炭素とアルコールをつくるのです。そのほかにも酵母や麹菌は、酢、味噌、醤油などの醸造食品に広く用いられています。私たちは、これら酵母や麹菌が、醸造における様々な環境に適応するメカニズムの解明を行うことでより良い発酵食品を作ることのできる醸造微生物の育種をめざして研究を行っています。
また、麹菌はデンプンを分解する酵素やクエン酸を多量に分泌生産するなど高い物質生産能力をもつことから、発酵食品にとどまらず様々な有用物質の生産宿主としても利用されています。私たちは、その能力の基礎となるメカニズムを解明し、有用物質の生産に応用する研究にも力を入れています。
以下に、当研究室の研究テーマをいくつか紹介します。なかにはすでに完了したものや、現在進行中のため詳細を公開できないものもあります。研究は状況に応じて柔軟に進めています。醸造微生物学の研究に興味のある方は、どうぞお気軽にお問い合わせください。
研究テーマ
- 焼酎酵母分子育種のための基盤形成
- 酵母の細胞膜リン脂質リモデリングの解明
- 酵母の栄養源認識とシグナル伝達経路の解明
- 麹造りにおける白麹菌のクエン酸高生産機構の解明
- 焼酎麹菌のクエン酸輸送体の解析
- ゲノム情報を活用した焼酎麹菌の解析
- 鰹節カビの分類と同定
- 鰹節カビの生活環と好乾性に関する研究
- 焼酎麹菌と鰹節カビの研究基盤の構築
①焼酎酵母分子育種のための基盤形成
酵母の育種においては、特定の遺伝子を働かなくすることで、これまでにない代謝生成物の蓄積により新たな味や香りの製品を生み出すことができ、その組み合わせは無限に存在すると考えられています。
これまでの酵母の育種法は、紫外線照射や変異剤処理により遺伝子に損傷を引き起こすことで行われてきました。しかしこの方法では、目的の遺伝子のみをターゲットとすることはできず、また目的以外の遺伝子にも損傷を与えてしまう恐れを含んでいるのです。さらに、醸造用酵母は2倍体であるために同じ遺伝子を2つ持つことから、それら2つの遺伝子を1度に働かなくすることは確率的に非常に低く、そのため変異株の取得には多大な時間と労力を要するものなのです。私たちは、ヘテロ接合性消失(LOH; Loss of Heterozygosity)という現象を利用した簡便な焼酎酵母の遺伝子破壊方法の構築を目的として研究を行っています。
また、交配による優良株の育種法の開発にも取り組んでいます。醸造用酵母は通常2倍体ですが、減数分裂後に生じた胞子が発芽した1倍体細胞は異なる接合型の株同士で接合して2倍体となります。このため異なる株同士の接合による育種が試みられてきました。しかしながら清酒酵母は胞子形成能ならびに胞子発芽能が極めて低いことから、これまで交配による育種法は確立されていません。私たちは、焼酎酵母が清酒酵母と近縁であるにもかかわらず高い胞子形成能ならびに胞子発芽能を有することを見出しました。また、ホモタリック注1)な焼酎酵母においても、マイクロマニピュレーターを用いて単離した胞子を隣接させることで確実に交配株が得られる顕微接合法を開発し(図1A)、交配による育種法についても研究しています。
注1)出芽酵母の1倍体細胞は2つの接合型(aとα)のいずれかを示す。ホモタリック株は、増殖に際し一部の細胞で接合型が変換するが(図1B)ヘテロタリック株では接合型変換は起こらない。
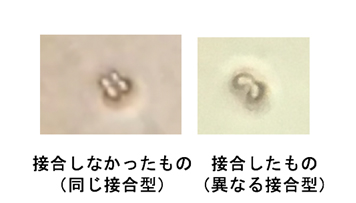
図1A 顕微接合法による焼酎酵母の接合実験

図1B ホモタリック株における接合型変換
②酵母の細胞膜リン脂質リモデリングの解明
細胞膜は半透性の脂質2重層からできていてこれによって細胞質と外環境を隔てています。細胞膜の主な構成成分であるリン脂質は、酸化などによって不飽和結合が切断された場合などに短くなったアシル鎖を切り離しリゾリン脂質とした後、新たなアシル鎖を導入するメンテナンス機構が存在し、これを脂質のリモデリングといいます(図2)。前者を触媒するのがホスホリパーゼなどのエステル分解酵素であり、後者を触媒するのがアシル転移酵素と考えられています。しかしながら、これらの酵素とリモデリングの関連はほとんど解明されておらず、特にリゾリン脂質を受容体基質とするアシル転移酵素に関しては全く同定されていませんでした。
私たちは、出芽酵母全遺伝子のうち致死でない約5,000の遺伝子を1つずつ破壊した遺伝子破壊株のセットについてスクリーニングを行い、1株だけ活性を持たない株を見出しLPT1と命名しました。LPT1は、membrane bound O-acyltransferase (MBOAT) スーパーファミリーのモチーフを持ち、酵母からヒトにいたるまでホモログが見出されましたがこれまでに研究されていない新規な酵素であることが分かりました。私たちは、Lpt1-GFPが小胞体膜上に局在すること(図3)、Lpt1がさまざまなリン脂質に基質特異性を示すこと、リゾフォスファチジン酸 (LPA)の生合成をもう一つのアシル転移酵素Slc1と協調して行うことを明らかにするとともにさらなる研究を続けています。
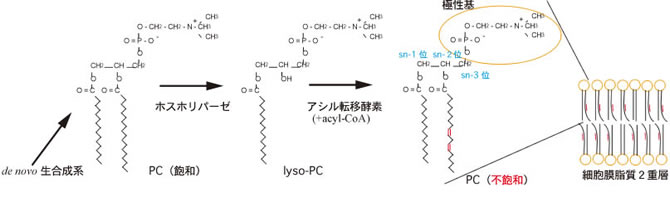
図2 リン脂質のリモデリング
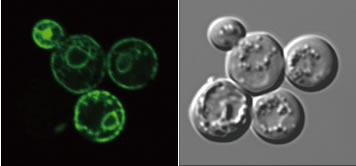
図3 LPT1-GFPの細胞内局在性
LPT1 encodes a membrane-bound O-acyltransferase involved in the acylation of lysophospholipids in the yeast Saccharomyces cerevisiae. Tamaki, H., Shimada, A., Ito, Y., Ohya, M., Takase, J., Miyashita, M., Miyagawa, H., Nozaki, H., Nakayama, R., and Kumagai, H. (2007) J. Biol. Chem., 282, 34288-34298.
③酵母の栄養源認識とシグナル伝達機構の解明
酵母は自らを取り巻く環境中の栄養状況をモニターして増殖や分化への切換えを行っています。たとえば代表的な栄養源であるグルコースは、細胞内のcAMPレベルを上昇させることで増殖のスイッチをONにすると考えられています。ところが、グルコースを感知する受容体に関しては、これまで全くわかっていませんでした。私たちは、グルコースシグナル伝達経路に関与すると考えられていた3量体Gタンパク質に着目し、その分子と共役する遺伝子GPR1をクローニングすることに成功しました。GPR1は7回膜貫通型の典型的なGタンパク質共役型受容体(図4,左上)をコードしており、クラゲの緑色蛍光タンパク質(GFP)と融合させて共焦点レーザー顕微鏡で観察すると、細胞表層の細胞膜に局在することがわかりました(図4,左下)。GPR1遺伝子を破壊した株では、グルコース添加による細胞内cAMPレベルの一過的な上昇が見られなくなったことから(図4,右上)Gpr1タンパク質は、グルコース受容体であることが明らかとなりました。出芽酵母は栄養状況に応じて形態を変化させます。たとえば2倍体細胞を高グルコース・低窒素源条件下におくと、偽菌糸形成 (Pseudohyphal growth) とよばれる一見カビの様な菌糸状の増殖形態をとります(図4,右下)。私たちは、GPR1遺伝子破壊株およびGタンパク質であるGPA2遺伝子破壊株では、偽菌糸形成が起こりにくくなり、また培地中へのcAMPの添加が偽菌糸形成不全を回復させる(抑圧する)ことからcAMP経路は細胞の増殖のみならず分化をも制御していることを明らかにしました。
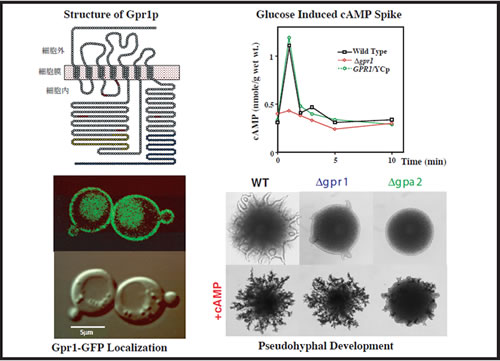
図4 出芽酵母のGpr1
- Loss of heterozygosity is induced in Candida albicans by ultraviolet irradiation. Takagi, Y., Akada, R., Kumagai, H., Yamamoto, K., and Tamaki, H. (2008) Appl. Microbiol. Biotechnol., 77, 1073-1082
- Glucose-Stimulated cAMP-PKA Pathway in Yeast Saccharomyces cerevisiae. Tamaki, H. (2007) J. Biotech. Bioeng., 104(4), 245-250.
- Glucose-dependent cell size is regulated by a G protein-coupled receptor system in yeast Saccharomyces cerevisiae. H. Tamaki, C.-W. Yun, T. Mizutani, T. Tsuzuki, Y. Takagi, M. Shinozaki, Y. Kodama, K. Shirahige, and H. Kumagai. (2005) Genes Cells, 10 (3), 193-206.
- Gpr1, a Putative G-Protein-Coupled Receptor, Regulates Morphogenesis and Hypha Formation in the Pathogenic Fungus Candida albicans. T. Miwa, Y. Takagi, M. Shinozaki, C.-W. Yun, W. A. Schell, J. R. Perfect, H. Kumagai, and H. Tamaki.(2004)Eukaryot. Cell, 3 (4), 919-931.
- Characterization of heterotrimeric G protein complexes in rice plasma membrane.Kato, T., Mizutani, T., Tamaki, H., Kumagai, H., Kamiya, T., Hirobe, A., Fujisawa, Y., Kato, H., and Iwasaki, Y. (2004) Plant J., 38 (2), 320-331.
- GPR1 regulates filamentous growth through FLO11 in yeast Saccharomyces cerevisiae. Tamaki, H., Miwa, T., Shinozaki, M., Saito M., Yun, C.-W., Yamamoto, K., and Kumagai, H. (2000) Biochem. Biophys. Res. Commun., 267, 164-168.
- Gpr1p, a putative G-protein coupled receptor, regulates glucose dependent cellular cAMP level in yeast Saccharomyces cerevisiae. Yun, C.-W., Tamaki, H., Nakayama, R., Yamamoto, K. and Kumagai, H. (1998) Biochem. Biophys. Res. Commun., 252, 29-33
- G-Protein Coupled Receptor from Yeast Saccharomyces cerevisiae. Yun, C.-W., Tamaki, H., Nakayama, R., Yamamoto, K., and Kumagai, H. (1997) Biochem. Biophys. Res. Commun., 240, 287-292
④麹造りにおける白麹菌のクエン酸高生産機構の解明
焼酎の製造において、白麹菌が生産するクエン酸は、もろみを酸性にして雑菌の生育を抑える重要な役割があります。麹を製造する工程において、前半は麹菌の増殖にともなう発酵熱により比較的高温で推移しますが、クエン酸は高温ではあまり生産されないため、後半では温度を低下させる必要があります(図5Aの実線)。しかし、温度を下げるとなぜクエン酸が生産されるのかについては分かっていませんでした。
私たちは、この温度管理とクエン酸生産との関係を明らかにするために、白麹を製造する際に、温度を低下させた場合とさせなかった場合の遺伝子発現の違いを網羅的に解析しました。その結果、白麹菌のゲノムにコードされている約1万2千の推定遺伝子のうち約1割の発現が異なることが示唆されました。そこで、それらの発現量の変化した遺伝子を解析したところ、温度を下げると解糖系から分岐するトレハロース合成経路、グリセロール合成経路、ペントースリン酸経路の代謝系遺伝子の発現レベルが減少することが分かりました(図5B)。このことから、温度が低下するとTCA回路へのカーボンフローが増強されてクエン酸生産が促進されると考えられました。また、トレハロースやグリセロールには各種ストレスに対して細胞を保護する機能が知られていることや、温度低下によって多くの熱ショックタンパク質をコードする遺伝子の発現レベルも減少することから、麹造りで行われる後半の温度低下の工程は、クエン酸生産を促進するというよりも、熱ストレス応答によるクエン酸生産の抑制から解放する意味があると考えられました。
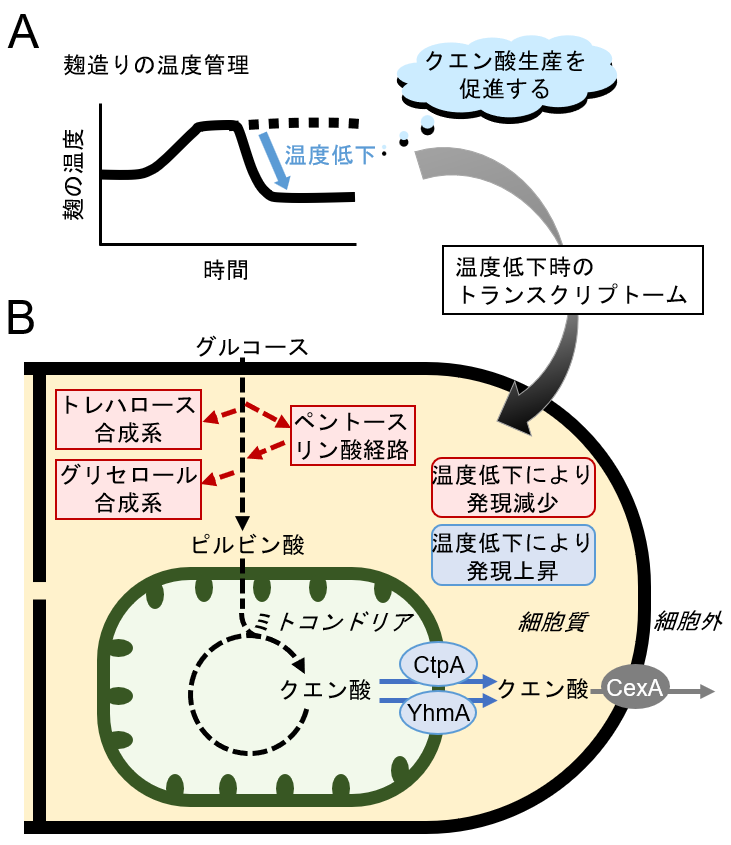
図5 麹造りの温度管理(A)と遺伝子発現の変化(B)
- 二神泰基, 後藤正利.白麹菌の研究ツールの開発とそれを利用したクエン酸高生産機構の解析. 科学と工業 96(3) 2022.
- Futagami T, Goto M. (2022) Insights regarding sirtuin-dependent gene regulation during white koji production. Commun Integr Biol. 15(1):92-95.
- 二神泰基, 玉置尚徳, 後藤正利, 高峯和則. 続・生物工学基礎講座 バイオよもやま話 焼酎学へのいざない. 生物工学会誌 97(2), 82-86. 2019.
- Futagami T, Mori K, Wada S, Ida H, Kajiwara Y, Takashita H, Tashiro K, Yamada O, Omori T, Kuhara S, Goto M. (2015) Transcriptomic analysis of temperature responses of Aspergillus kawachii during barley koji production. Appl. Environ. Microbiol. 81, 1353-1363.
- Futagami T, Mori K, Yamashita A, Wada S, Kajiwara Y, Takashita H, Omori T, Takegawa K, Tashiro K, Kuhara S, Goto M. (2011) Genome sequence of the white Koji mold Aspergillus kawachii IFO 4308 used for brewing the Japanese distilled spirit Shochu. Eukaryot. Cell. 10, 1586-1587.
⑤焼酎麹菌のクエン酸輸送体の解析
焼酎製造に使用される白麹菌や黒麹菌が、なぜクエン酸を高レベルで分泌生産できるのか、その仕組みはこれまで明らかになっていませんでした。私たちは、クエン酸高分泌生産機構の解明のため、クエン酸輸送体について解析を行いました。
クエン酸は、ミトコンドリアのTCA(Tricarboxylic Acid)回路の中間代謝物として合成されたものが、細胞質へと輸送され、さらに細胞外へと排出されます。そこで、まずクエン酸がミトコンドリアから細胞質へと輸送されるために必要なクエン酸輸送体について解析し、ミトコンドリア膜ではCtpAとYhmAが主要なクエン酸輸送体であることを明らかにしました(図6)。また、CtpAとYhmAによって細胞質に排出されたクエン酸の一部は、麹菌が生存するために必須なアセチルCoAの合成に使われていることを明らかにしました。次に、細胞質膜に局在するクエン酸輸送体CexAがクエン酸高生産にきわめて重要であることを明らかにしました。たとえば、白麹菌のクエン酸輸送体CexAを、黄麹菌に導入すると、黄麹菌が白麹菌に匹敵するクエン酸分泌生産能力を獲得することが示されました(図7)。なお、白麹菌においてcexA遺伝子の発現がエピジェネティックに制御されることも分かったため、その発現制御機構を解明のために、さらなる研究を続けています。これらのクエン酸輸送体に関する知見は、有用物質生産における輸送体をターゲットとした育種戦略への活用が期待されます。

図6 白麹菌におけるクエン酸排出モデル

図7 黄麹菌において白麹菌のクエン酸輸送体CexAを発現させるとクエン酸を高分泌するようになる。培地にはpH指示薬としてメチルレッドを添加しており、CexA発現株のコロニーは周囲のpHが下がるため赤いハローをつくる。
- Kadooka C, Izumitsu K, Asai T, Hiramatsu K, Mori K, Okutsu K, Yoshizaki Y, Takamine K, Goto M, Tamaki H, Futagami T. (2025) Overexpression of the RNA-binding protein NrdA affects global gene expression and secondary metabolism in Aspergillus species. mSphere.10(2):e0084924
- Nishitani A, Hiramatsu K, Kadooka C, Mori K, Okutsu K, Yoshizaki Y, Takamine K, Tashiro K, Goto M, Tamaki H, Futagami T. (2023) Expression of heterochromatin protein 1 affects citric acid production in Aspergillus luchuensis mut. kawachii. J Biosci Bioeng. 136(6):443-451.
- 二神 泰基, 門岡 千尋, 後藤 正利, 玉置 尚徳. 白麹菌のクエン酸高生産機構ークエン酸生産能力の鍵をにぎる輸送体. 化学と生物 59(9), 241-246. 2021.
- 二神 泰基, 門岡 千尋, 後藤 正利, 玉置 尚徳. 麹菌のクエン酸高生産機構. 日本醸造協会誌 116(12), 810-818. 2021.
- Nakamura E, Kadooka C, Okutsu K, Yoshizaki Y, Takamine K, Goto M, Tamaki H, Futagami T. (2020) Citrate exporter enhances both extracellular and intracellular citric acid accumulation in the koji fungi Aspergillus luchuensis mut. kawachii and Aspergillus oryzae. J. Biosci. Bioeng. 2021 131(1):68-76.
- 二神泰基, 門岡千尋. 焼酎造りに用いられる白麹菌のクエン酸高生産機構. バイオサイエンスとインダストリー. 78, 213-216, 2020.
- Kadooka C, Nakamura E, Mori K, Okutsu K, Yoshizaki Y, Takamine K, Goto M, Tamaki H, Futagami T. (2020) LaeA controls citric acid production through regulation of the citrate exporter-encoding cexA gene in Aspergillus luchuensis mut. kawachii. Appl Environ Microbiol. 86, e01950-19.
- Miyamoto A, Kadooka C, Mori K, Tagawa Y, Okutsu K, Yoshizaki Y, Takamine K, Goto M, Tamaki H, Futagami T. (2020) Sirtuin SirD is involved in α-amylase activity and citric acid production in Aspergillus luchuensis mut. kawachii during a solid-state fermentation process. J Biosci Bioeng. 129, 454-466.
- Kadooka C, Izumitsu K, Onoue M, Okutsu K, Yoshizaki Y, Takamine K, Goto M, Tamaki H, Futagami T. (2019) Mitochondrial citrate transporters CtpA and YhmA are required for extracellular citric acid accumulation and contribute to cytosolic acetyl-CoA generation in Aspergillus luchuensis mut. kawachii. Appl. Environ. Microbiol. 85, AEM.03136-18.
⑥ゲノム情報を活用した焼酎麹菌の解析
近年の生物学における研究において、ゲノム配列や遺伝子発現に基づく解析は不可欠な手法となっています。私たちは、白麹菌 IFO(NBRC)4308株や黒麹菌RIB2601株のゲノムを解析し、8本の染色体と1本のミトコンドリアにまとまったゲノム情報を取得しています。
このゲノム情報をもとに、それぞれの微生物に特徴的な表現型や性質の解明にも取り組んでいます。たとえば、白麹菌のゲノム解析の結果、その白色化の要因が黒色のメラニン色素の合成に関与するpksP遺伝子の破壊的変異であることが示唆されました。実際に、白麹菌のpksP遺伝子の変異を修復することで黒麹菌のように黒色胞子を形成するようになったことから、pksPの変異が白色化の要因であることが明らかになりました(図8)。
また、私たちは白麹菌IFO4308株のゲノムにコードされるβ-キシロシダーゼ遺伝子に変異が入っていることに気が付きました。β-キシロシダーゼはキシロビオースなどのキシロオリゴ糖の分解に関わる酵素で、その産物としてキシロースが生じます(図9)。キシロースは、焼酎の蒸留過程において、クエン酸による低pH環境下で加熱されることにより、香気成分であるフルフラールへと変換されることが知られています。実際に、IFO4308株を用いて製造した焼酎のもろみでは、キシロースの含量が低いことが確認されました。このように私たちは、ゲノム情報に基づいた解析を通じて、それぞれの焼酎麹菌の特徴の理解を深める研究を進めています。
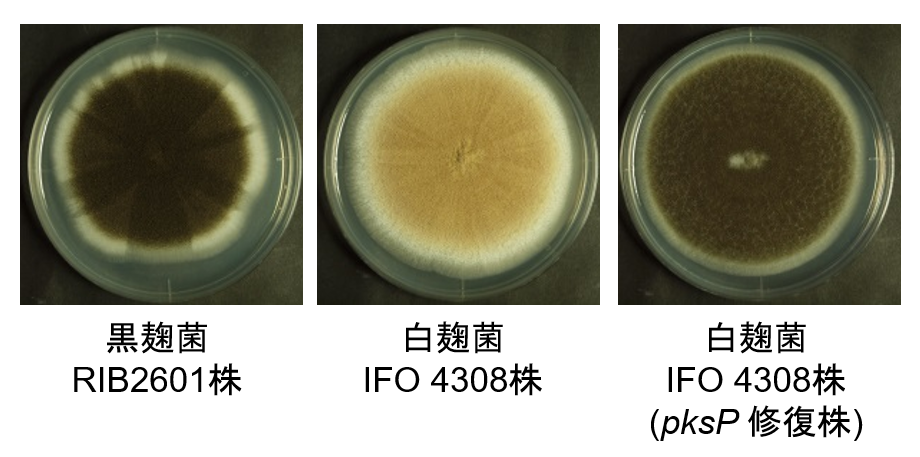
図8 白麹菌においてpksPの変異を修復すると黒色の分生子を形成する。
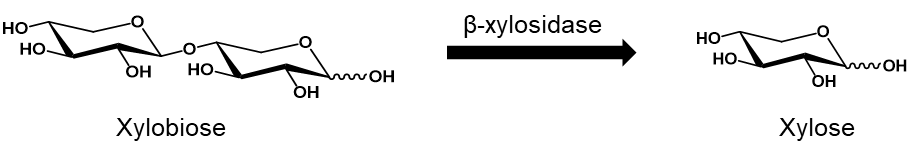
図9 麹菌が生産するβ-キシロシダーゼ
- 西谷篤, 二神泰基. 白麹菌における新規な有機酸排出トランスポーターの探索. アグリバイオ 8(4) 2024.
- 二神泰基, 玉置尚徳. お酒の生物学[日本編] 焼酎造りを支える麹菌と酵母の新展開-遺伝子から特徴的性質を紐解く. 生物の科学 遺伝. 75(3) 2021.
- Zhu E, Hiramatsu K, Inoue T, Mori K, Tashiro K, Fujita K, Karashima T, Takashita H, Okutsu K, Yoshizaki Y, Takamine K, Tamaki H, Futagami T. (2024) Deficiency of β-xylosidase activity in Aspergillus luchuensis mut. kawachii IFO 4308. Biosci Biotechnol Biochem. 88(7):816-823.
- Nishitani A, Hiramatsu K, Kadooka C, Hiroshima K, Sawada K, Okutsu K, Yoshizaki Y, Takamine K, Goto M, Tamaki H, Futagami T. (2024) Overexpression of the DHA1 family, ChlH and ChlK, leads to enhanced dicarboxylic acids production in koji fungi, Aspergillus luchuensis mut. kawachii and Aspergillus oryzae. J Biosci Bioeng. 137(4):281-289.
- Futagami T. (2022) The white koji fungus Aspergillus luchuensis mut. kawachii. Biosci Biotechnol Biochem. 86(5):574-584.
- Mori K, Kadooka C, Oda K, Okutsu K, Yoshizaki Y, Takamine K, Tashiro K, Goto M, Tamaki H, Futagami T. (2022) Chromosome-level genome sequence data and analysis of the white koji fungus, Aspergillus luchuensis mut. kawachii IFO 4308. Data Brief.41:107888.
- Mori K, Kadooka C, Nishitani A, Okutsu K, Yoshizaki Y, Takamine K, Tashiro K, Goto M, Tamaki H, Futagami T. (2021) Chromosome-Level Genome Sequence of the Black Koji Fungus Aspergillus luchuensis RIB2601. Microbiol Resour Announc. 10(29):e0038421.
⑦鰹節カビの分類と同定
鰹節には、鰹を煮熟、焙乾をして製造される荒節と、そこからさらにカビ付けと天日干しを繰り返して製造される本枯節に分けられます。本枯節はカビが生産する脂質およびタンパク質の分解酵素の作用によって、風味はまろやかに、色合いは上質なものになります。カビ付けの方法は製造会社によって異なっており、販売されている優良黴菌を使用している製造会社が多いですが、湿度と温度が管理されたカビ付け室で自然に生えてくるカビを用いる、昔ながらの方法でカビ付けを行う会社もあります。
そこで、私たちはカビ付け室の自然発生カビにより製造された本枯節に着目し、その菌叢を調査しました。その結果、Aspergillus chevalieri、Aspergillus montevidensis、Aspergillus sydowiiなどが存在することを明らかにしました。そのなかでもA. chevarieriが1番カビから3番カビにかけて50%~85%を占めており、主要な存在だと考えられました。また、同じA. chevalieriであるにもかかわらず異なる表現型を示す2つの株が同定されました。これらの菌株は、カルモジュリンやβ-チューブリン遺伝子によるDNAを用いた系統解析の結果ではどちらもA. chevalieriに分類されるが、有性生殖により子嚢胞子をつくる有性世代株と無性生殖により分生子をつくる無性世代株に分類された。すなわち、A. chevalieriには有性世代と無性世代の2つの生活環を有することが分かりました。
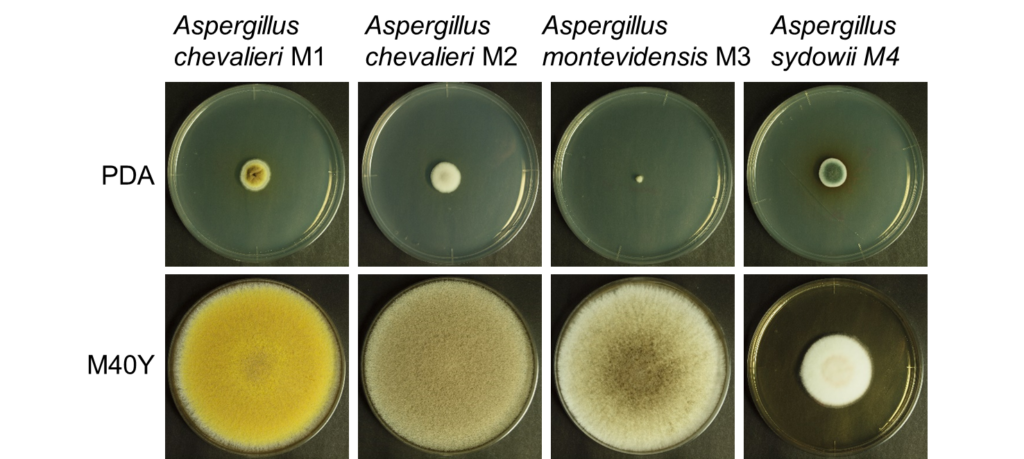
図10 枯節から分離された鰹節カビの同定
- Kadooka C, Nakamura E, Kubo S, Okutsu K, Yoshizaki Y, Takamine K, Tamaki H, Futagami T. (2020) Analysis of the fungal population involved in Katsuobushi production. J. Gen. Appl. Microbiol. 66(4):239-243.
⑧鰹節カビの生活環と好乾性に関する研究
私たちは、鰹節カビ Aspergillus chevalieri に、有性世代と無性世代の両方が存在することを明らかにしました。Aspergillus 属の糸状菌には、有性世代と無性世代の両方の生活環を持つ種と、有性生殖能力を失い無性世代のみで生活する種が存在します。たとえば、酒・味噌・醤油の製造に使用される麹菌では、これまでに有性世代の存在は確認されておらず、無性世代のみであると考えられています。このような背景から、私たちは鰹節カビの有性生殖能力に興味をもっています。
しかし、麹菌と比較すると、鰹節カビに関する遺伝子レベルの研究はほとんど進んでいないのが現状です。そこで私たちはまず、鰹節から単離された A. chevalieri の有性世代株についてゲノム解析を行いました。その結果、この株は接合型決定因子である MAT1-1-1 および MAT1-2-1 の両方を有しており、表現型のみならず遺伝的背景からも、本菌が自家不和合性を示さないホモタリック型(雌雄同体性)であることが裏付けられました。現在、私たちは鰹節カビの生活環や、高い乾燥耐性(好乾性)といった特徴的な生命現象の解明に向けた研究を進めています。

図11 鰹節カビAspergillus chevalieriの子嚢胞子(有性胞子)と分生子(無性胞子)の形成
- Kadooka C, Mori K, Okutsu K, Yoshizaki Y, Takamine K, Tashiro K, Tamaki H, Futagami T. (2021) Chromosome-Level Genome Sequence of Aspergillus chevalieri M1, Isolated from Katsuobushi. Microbiol Resour Announc. 10(37):e0038521.
- 門岡 千尋, 合原 翔平, 久保 臣悟, 尾上 昌平, 奥津 果優, 吉﨑 由美子, 髙峯 和則, 後藤 正利, 玉置 尚徳, 二神 泰基. (2020) 白麹菌Aspergillus luchuensis mut. kawachii NBRC 4308のアルビノ表現型の原因遺伝子の解析.日本醸造協会誌.115(6) 369-377.
⑨焼酎麹菌と鰹節カビの研究基盤の構築
近年、DNAシーケンス技術や質量分析技術の進展により、ゲノム情報や網羅的な遺伝子・タンパク質の発現情報を容易に取得できるようになりました。私たちも、麹菌によるクエン酸や酵素の高効率な細胞外分泌のメカニズムや、鰹節カビの生活環の変化といった現象を解析するために、これらの先端技術を活用しています。しかし、こうした膨大なオミクス情報に基づく予測を実験的に検証するには、遺伝子組換えやゲノム編集などの遺伝子操作技術が不可欠です。私たちは、麹菌および鰹節カビを対象としたこれらの技術の開発にも取り組んでいます。たとえば、目的のゲノム領域に遺伝子を効率的に導入してその機能を喪失させるノックアウトや、緑色蛍光タンパク質(GFP)を融合させたタンパク質を発現させて局在や発現動態を可視化する手法などを確立し、遺伝子機能の解明に応用しています。
麹菌や鰹節カビを含む糸状菌のおおくは、損傷したDNAを修復する際に非相同末端結合(Non-Homologous End Joining, NHEJ)という修復機構が活発にはたらくために目的の部位で遺伝子組換えすることがむずかしいという問題があります。そこで、NHEJに関わる遺伝子をあらかじめ破壊することで相同組換えを効率化することが行われています。私たちは、白麹菌、黒麹菌と鰹節カビにおいて非相同末端結合に必要なligD遺伝子やku70遺伝子を破壊することにより相同組換え効率が劇的に上昇した宿主を構築して、それらを研究に用いています。また、ゲノム編集技術であるCRISPR-Cas9システムを用いて、非相同末端結合が正常にはたらいている白麹菌の野生株においても高効率で遺伝子をノックアウトする技術を開発しました(図12)。現在も、麹菌や鰹節カビの研究開発を促進するための基盤整備を続けています。
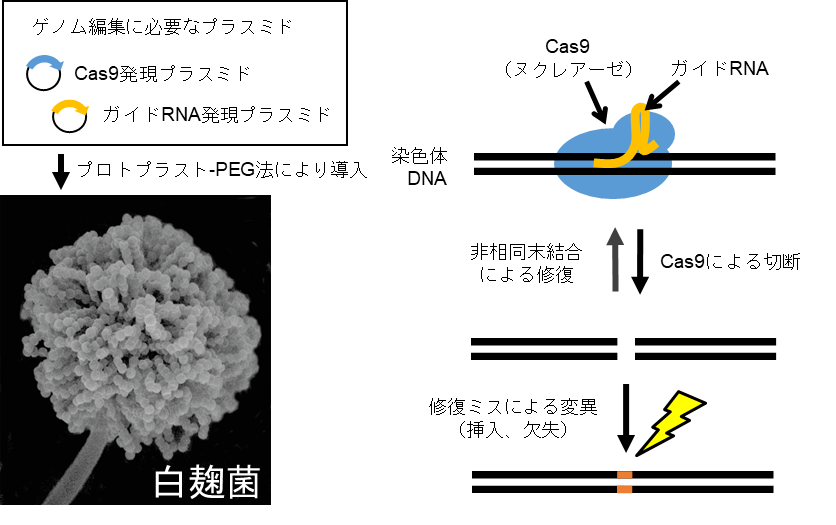
図12 白麹菌におけるゲノム編集系の概要
- 平松健太郎, 二神泰基. 鰹節カビAspergillus chevalieriの研究基盤. アグリバイオ 7(8) 2023.
- Hiramatsu K, Nishitani A, Okutsu K, Yoshizaki Y, Takamine K, Tamaki H, Futagami T. (2023) Efficient gene targeting in Aspergillus chevalieri used to produce katsuobushi. Biosci Biotechnol Biochem. 87(6):672-682.
- Chromosome-Level Genome Sequence of Aspergillus puulaauensis MK2, a Fungus Isolated from a Dead Hard Tick. (2021) Futagami T, Mori K, Kadooka C, Niihara H, Tashiro K, Tamaki H, Tanaka T. Microbiol Resour Announc. 10(36):e0037221.
- Kadooka C, Yamaguchi M, Okutsu K, Yoshizaki Y, Takamine K, Katayama T, Maruyama JI, Tamaki H, Futagami T. (2020) A CRISPR/Cas9-mediated gene knockout system in Aspergillus luchuensis mut. kawachii. Biosci. Biotechnol. Biochem.84(10):2179-2183.
- Kimoto D, Kadooka C, Saenrungrot P, Okutsu K, Yoshizaki Y, Takamine K, Goto M, Tamaki H, Futagami T. (2019) Pex16 is involved in peroxisome and Woronin body formation in the white koji fungus, Aspergillus luchuensis mut. kawachii. J. Biosci. Bioeng. 127, 85-92.
- Kojo T, Kadooka C, Komohara M, Onitsuka S, Tanimura M, Muroi Y, Kurazono S, Shiraishi Y, Oda K, Iwashita K, Onoue M, Okutsu K, Yoshizaki Y, Takamine K, Futagami T, Mori K, Tamaki H. (2018) Characterization of amylolytic enzyme overproducing mutant of Aspergillus luchuensis obtained by ion beam mutagenesis. J. Gen. Appl. Microbiol. 63, 339-346.
- Kadooka C, Onitsuka S, Uzawa M, Tashiro S, Kajiwara Y, Takashita H, Okutsu K, Yoshizaki Y, Takamine K, Goto M, Tamaki H, Futagami T. (2016) Marker recycling system using the sC gene in the white koji mold, Aspergillus luchuensis mut. kawachii. J. Gen. Appl. Microbiol. 62, 160-163.
- 二神泰基, 後藤正利. 焼酎製造に用いられる白麹菌のゲノム解析と育種基盤の開発. バイオサイエンスとインダストリー. 74, 402-205, 2016.
